研究为肺癌治疗揭示新方向
肺腺癌是香港癌症死亡个案的主因之一,虽其发病成因复杂,但很多研究显示,RNA转录组的变动对癌症的发展有明显影响。有见及此,生命科学学院陈廷峰教授领导的研究团队开发了一种新RNA计算方法LAFITE (Low-abundance Aware Full-length Isoform clusTEr),成功在肺腺癌细胞系中识别出数千个现有技术无法捕获的低丰度全长 RNA 转录本,并从肺腺癌驱动基因中,识别出一种与促进癌细胞转移、患者存活率相关的稀有RNA转录本亚型,有助科学界了解肺癌形成、转移和进展的潜在机制。详情已发表于国际权威期刊Advanced Science。

生物体中的不同组织会共享相同的基因组,但它们在转录组(由不同表达水平的RNA转录本组成)方面存在显著差异。过往研究表明,大多数转录本的表达处于低水平(即低丰度),但它们在各种生物过程中具重要调节作用,包括代谢过程和癌症形成等。
RNA测序是一种广泛应用于生物学和临床研究的短读测序技术,方法是将RNA分子打碎后大量复制,再排序重组成更大的转录本。然而,它会倾向大量复制活跃的短RNA转录本令其易于捕获,结果低丰度的全长 RNA转录本因数量比例变小而难以捕获,因此RNA测序不擅于识别低丰度的全长转录本。
高等真核生物如人类的基因都存在转录本亚型,透过RNA剪接技术,可从同一基因产生多种转录本亚型。RNA测序的另一限制是无法检测单个转录本亚型,所以过往科学家大多研究转录组的基因水平,而非转录本水平,令大量低丰度的转录本未被发现。
虽然第三代测序技术「Oxford Nanopore Technologies」可以捕获天然 RNA 转录本,在识别低丰度的全长RNA转录本方面较为优胜,然而其产生的Nanopore DRS数据杂乱、内在错误多,容易影响数据的准确性。
陈教授的团队获香港研究资助局卓越领域计划和合作研究基金支持,运用Nanopore DRS数据进行转录本测序的新计算方法,开发了 LAFITE。它对低丰度的RNA 转录本具高灵敏度,可捕获全长转录本,与同类计算方法相比,表现更为优秀。
团队利用 LAFITE 研究了四种肺腺癌细胞系的Nanopore DRS 数据,成功从癌驱动基因AKT1中识别出一个新型低丰度的全长 RNA 转录本亚型,并证明它与促进癌细胞转移和患者存活率有关。
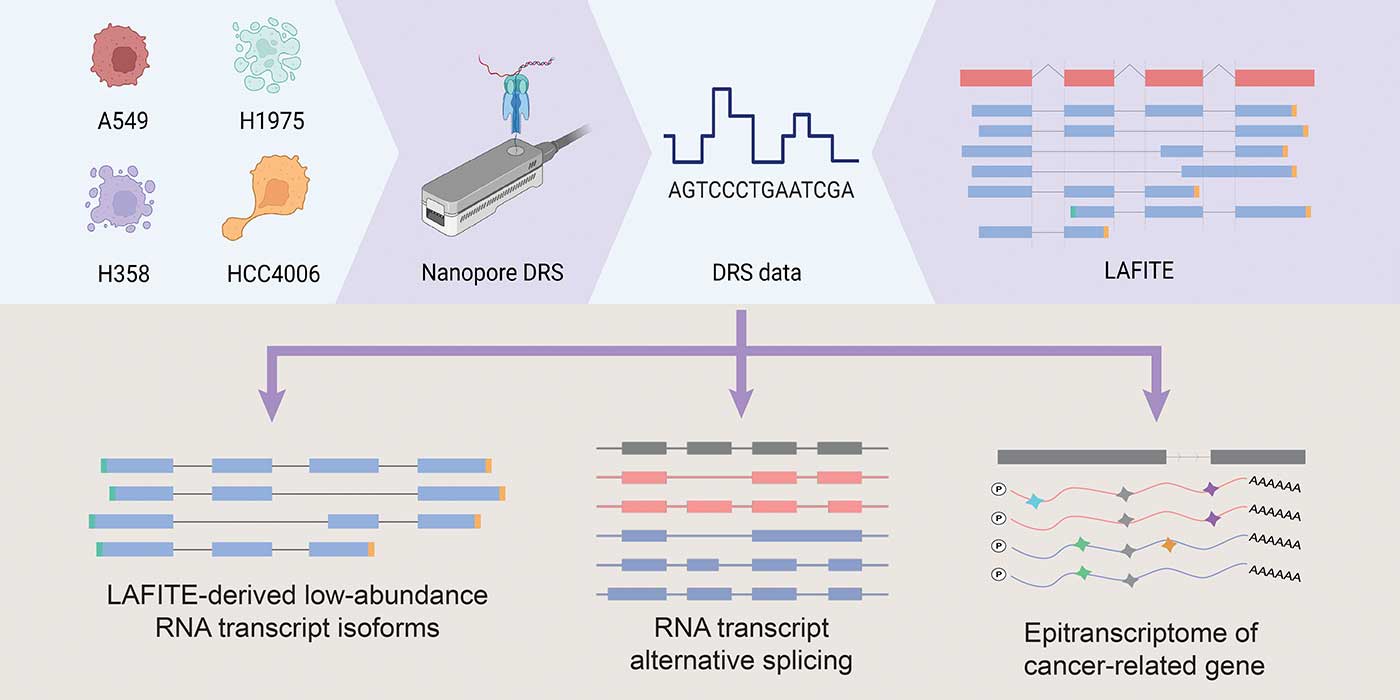
另外,团队从肺腺癌细胞系中识别出数千个未被发现的低丰度转录本,为肺腺癌研究提供更完整的全长RNA转录组,有助科学界了解与肺癌形成、转移和进展相关的潜在机制。新技术更有望应用于研究其他癌症,如结直肠癌,以及癌细胞的耐药性,促进药物研发。
陈教授表示﹕「如能对单个基因的所有转录本亚型进行全面研究,将有助了解其在生物学的功能。LAFITE能识别高及低表达水平的转录本,为科学界提供了评估基因功能的新方案,亦证明了从转录本水平层面研究转录组学的重要性。」
足本研究文章,可于此处阅览。